Le Deep Learning pour accélérer le diagnostic par imagerie médicale

Les avancés de l’Intelligence Artificielle sont vouées à bouleverser le monde de la santé. Nous présentons ici un exemple d’application du Deep Learning en imagerie médicale dans le cadre du Challenge Kaggle Data Science Bowl 2018.
Depuis 2014, le populaire challenge kaggle Data Science Bowl (DSB) a lieu tous les ans autour d’un défi de la santé. Pour l’année 2018 le Data Science Bowl a réuni de nombreux Data Scientist du monde entier pour s’attaquer à un des plus grands défis pour les biologistes et médecins : concevoir un algorithme de segmentation d’image pour automatiser la détection des noyaux dans les cellules biologiques.
Ce n’est pas par hasard que la segmentation est devenue un sujet d’actualité dans le traitement des images médicales.
La plupart des 30 milliards de cellules du corps humain contient un noyau rempli d’ADN, le code génétique qui programme chaque cellule. L’identification des noyaux des cellules est donc un point de départ important pour de nombreuses analyses médicales. L’identification des noyaux permet aux chercheurs d’isoler chaque cellule individuelle d’un échantillon, et suivre leur réaction à différents traitements. Ainsi, les chercheurs et les médecins peuvent comprendre les processus biologiques sous-jacents et accélérer le diagnostic médical.
Imaginez accélérer la recherche et le développement pour un grand nombre de maladies, du cancer du poumon aux maladies rares en passant par les maladies cardiaques. Chaque année des milliers de personnes en France et des millions dans le monde pourraient être sauvées s’ils avaient accès à un diagnostic plus rapide et précis de leur maladie.
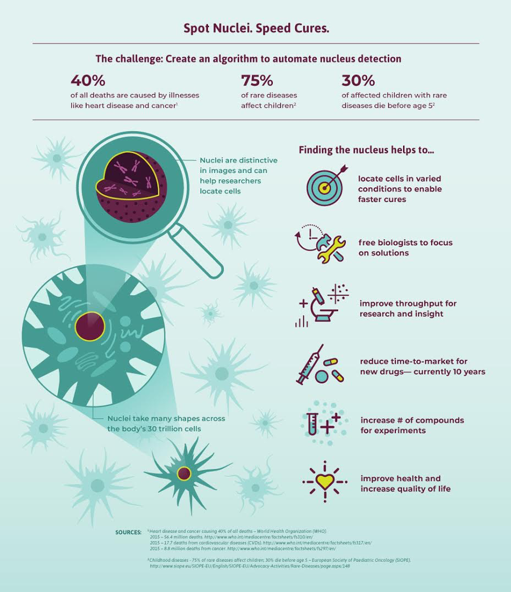
Figure 1 : Challenge Kaggle DSB 2018, Objectifs et motivations. Source : https://www.kaggle.com/c/data-science-bowl-2018
A Quantmetry, impossible pour nous de manquer un tel challenge. Nous avons donc constitué une équipe de Data scientist pour y participer.
Présentation du DSB 2018
1.Objectif
Il s’agit d’un problème de segmentation d’image difficile car il est question de détecter des objets parfois très peu semblables sur des fonds différents, avec le risque de confondre plusieurs objets (noyaux) distincts avec un unique objet. Nous verrons par la suite comment ce problème a pu être résolu par les approches mises en place.
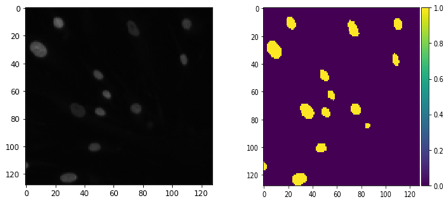
Figure 2 : Exemple d’image d’entrée pour le model (à gauche) et de masque d’apprentissage (à droite)
2. Caractéristiques du jeu de données
Une difficulté liée aux données réside dans l’hétérogénéité des images. En effet, les images sont très variées, présentent différents grossissements, différentes colorisations des noyaux et contiennent différents types cellulaires, comme on peut le voir sur la figure 3. De plus, nous disposons pour ce challenge de relativement peu de données : 673 images d’entraînement avec une majorité dominante (60%) pour les images à fond noir (cf figure 3 droite).
Plusieurs stratégies vont donc s’imposer pour pallier ces déséquilibres et cette taille de jeu de donnée.
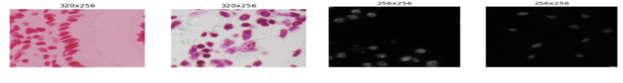
Figure 3 : Exemple d’images du Dataset kaggle du DSB 2018.
3. Métrique d’évaluation
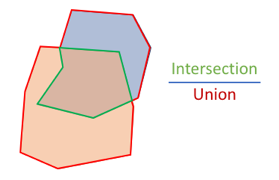
La métrique proposée par Kaggle est intéressante, car elle répond à une préoccupation double : nous souhaitons à la fois détecter et compter les cellules, mais aussi en déterminer le contour précis. Pour cela, la détection d’un noyau est principalement fondée sur un calcul d’IoU (Intersection over Union), une métrique classique en traitement d’image. On
dispose ainsi d’un indicateur permettant de déterminer si la prédiction recouvre pertinemment la ground truth. En choisissant un seuil sur cet IoU, on peut ainsi déterminer si un masque détecté correspond effectivement à un noyau ou non. On peut alors calculer les taux de vrais positifs, de faux positifs, de faux négatifs, et toute autre métrique classique en apprentissage.
La métrique retenue par Kaggle est un mélange entre précision et rappel, qui est ensuite moyenné pour différents seuils d’IoU (de 0.5 à 0.95). On obtient finalement le score par l’opération :
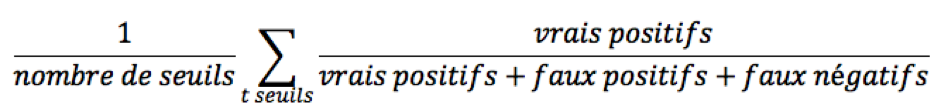
Quelle infrastructure pour ce challenge ?
Pour réaliser nos calculs nous utilisons un serveur de calcul Qbox de Quantmetry, une belle machine avec :
GPU NVIDIA 970 Ti
Mémoire vidéo : 8 GB GDDR5
- Intel socket 16 coeurs hyperthreadés @ 2.4GHz
- 64 Go RAM
- 2 x 1To SSD OS + Programmes
- 4To RAID10 ; 4 HDD 7200tr/min
- OS : Ubuntu 16
- Environnement : Python 3.6; Keras; Tensorflow
1. Petit échauffement : l’histogramme des couleurs
Nous commençons doucement afin de défricher le terrain en partant sur une stratégie simple mais efficace : l’histogramme des couleurs.
L’idée s’appuie sur l’observation que les images ont souvent un fond de couleur assez uniforme et que les noyaux se distinguent par une couleur différente. Ainsi l’idée est de dériver une répartition des niveaux de gris dans l’image et de définir la séparation entre les pixels des deux catégories de niveaux de gris. La catégorie des pixels de fort niveau de gris et la catégorie des faibles niveaux de gris peuvent correspondre aux noyaux et au background (fond) ou inversement selon le type d’image (cf figure 4).
Dans un premier temps les images sont converties en nuances de gris. Ainsi une image RGB de format : 256x256x3 est convertie en 256x256x1. Puis la médiane des pixels est calculée pour déterminer le seuil entre les deux catégories (background et noyaux)
Pour déterminer l’appartenance des pixels “background”, on regarde la catégorie des pixels majoritaires en supposant que les pixels du background sont majoritaires sur l’image. Dans les images à background sombre les pixels “clairs” seront donc classifiés “noyaux” et dans les images à background clair ce sont les pixels “sombres” qui seront classifiés “noyaux”.
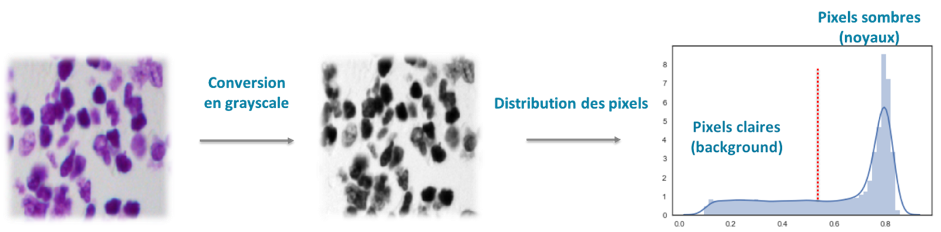
Figure 4 : Stratégie de l’histogramme des couleurs.
Cette stratégie nous a permis d’obtenir un score à 0.10 et d’être classé Top 78%. Elle fonctionne relativement bien sur les images à fond noir et noyaux clairs (cf Figure 5 – Haut gauche). En revanche elle pose plusieurs problèmes, le premier étant qu’elle est très sensible au bruit de fond dans l’image (cf Figure 5 – Bas gauche et Haut droite).
Le deuxième problème est la mauvaise segmentation des différents noyaux qui se touchent ou sont proches dans l’espace créant ainsi des agglomérats de noyaux (cf Figure 5 – Bas gauche et Haut droite).
Si cette approche naïve et rapide permet facilement d’obtenir des premiers résultats, elle se heurte bien vite à la complexité des données, que ce soit les nuances au sein d’une image ou leur diversité à travers l’ensemble du dataset.

Figure 5 : Quelques résultats de la stratégie ‘histogramme des couleurs’ ainsi que les deux types de problèmes rencontrés avec cette stratégie.
Après ce petit échauffement nous avons décidé de passer aux choses sérieuses, et d’utiliser des algorithmes à base de CNN, devenus une référence en traitement d’image depuis plusieurs années.
2. Un CNN, qu’est ce que c’est ?
Un CNN, c’est avant tout un réseau de neurone. Une donnée d’entrée, en l’occurrence une image, parcourt les différentes couches du réseau qui apprend à en extraire des caractéristiques, les features, qui représentent la donnée d’entrée à un niveau sémantique de plus en plus haut, jusqu’à permettre de pouvoir par exemple la classifier avec une probabilité associée : « cette image représente un camion », ou « un arbre » ou encore « un data scientist en milieu extérieur » (rare).
La particularité d’un réseau de neurone à convolution vient en fait de l’hypothèse d’invariance spatiale des caractéristiques utilisées dans l’image : on s’intéresse aux mêmes motifs à reconnaître dans les différentes parties de l’image. Techniquement, cela revient à faire partager à l’ensemble des neurones d’une même couche des poids similaires, ce qui permet de réduire considérablement le nombre de paramètres du réseau. A noter que cette hypothèse peut néanmoins limiter l’exploitation de structures bien spécifiques dans une image, comme la géométrie d’un visage Pour une bonne illustration plus détaillée du fonctionnement des étapes de convolutions, c’est ici.
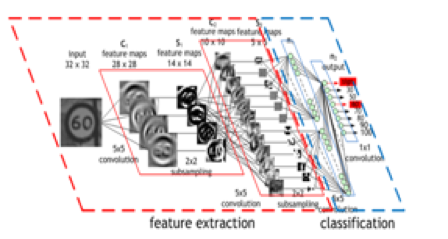
Figure 6 : Exemple de fonctionnement d’un CNN sur la reconnaissance de panneaux de signalisation. source : developer.nvidia.com
3. La piste U-Net
Maintenant que nous avons rappelé le principe du CNN, nous allons présenter le U-Net, premier bon candidat pour notre cas d’application. Le U-Net est une architecture à réseau convolutionnel pour une segmentation rapide et précise des images (cf figure 6). Il a la particularité d’avoir une architecture symétrique composée d’une partie de “contraction”, qui va permettre de détecter le contexte et les objets, et une partie “expansion” composée de succession d’up-convolution qui fait le chemin inverse qui va permettre de reconstituer l’image composée de macro-features extraites de la partie “contraction” et finalement de localiser précisément le contour des objets détectés.
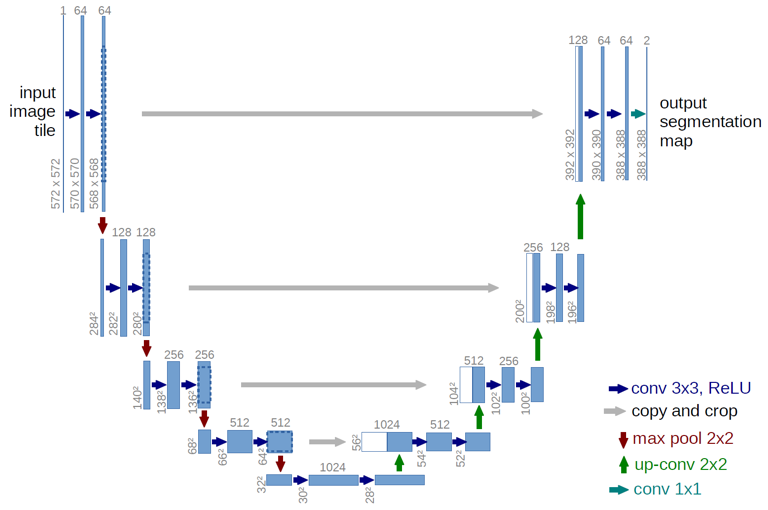
Figure 6 : Architecture du réseau U-Net.
Il est particulièrement adapté à la segmentation dans les images médicales (microscopes et radiographies). Jusqu’à présent, il a surpassé la meilleure méthode antérieure (un réseau convolutionnel à fenêtre glissante) sur le défi de l’ISBI pour la segmentation des structures neuronales dans les empilements microscopiques électroniques. Il a remporté le Grand Défi de la détection automatisée des caries en radiographie bitewing à l’ISBI 2015, et il a remporté le Défi du suivi cellulaire à l’ISBI 2015 sur les deux catégories de microscopie à lumière transmise. Il a également l’avantage de fonctionner sur des jeu de données de moyenne à petite taille (une trentaine d’image peut suffir) et se révèle rapide à entraîner.
En utilisant le U-Net tel quel, nous parvenons à un score de 0.32 et à atteindre le top 39%. Dans notre cas, le désavantage du U-net est qu’il produit une segmentation à une seule classe, en un seul masque (c.a.d une probabilité d’appartenir à un noyaux sur toute l’image). Pour dissocier les noyaux entre eux, la sortie du U-Net nécessite un vrai travail de post-traitement. Avant ce post-traitement, on considère par défaut comme un noyau les ensembles de pixels adjacents classifiés comme tels, mais qui peuvent correspondre à deux noyaux collés.
Par la suite nous avons donc appliqué du post-traitement visant à séparer artificiellement les noyaux proches dans l’espace sur la sortie du réseau U-Net.

Figure 8 : Post-traitement effectué sur le sortie du U-net visant à séparer les noyaux superposés.
Une approche simple consiste notamment à utiliser une hypothèse de convexité sur les noyaux. Si cette hypothèse est raisonnable et permet de séparer un bon nombre de noyau, elle n’est pas non plus toujours vérifiée.
En effet, la plupart des noyaux ont une forme convexe, même s’il peut arriver qu’ils présentent des défauts de convexité, comme le petit noyau de la figure 8. On constate cependant que le défaut de convexité de l’union des deux noyaux est largement plus prononcé. On considère donc que les défauts de convexité les plus importants correspondent en fait à une zone où deux noyaux sont en contact.
On commence ainsi par extrapoler la forme convexe la plus proche de la forme à séparer, qui constitue une sorte d’enveloppe. On cherche ensuite les points de notre forme les plus distants de cette extrapolation convexe, qui correspondent potentiellement à des séparations entre noyaux. Après seuillage sur des distances suffisamment grandes (renfoncement important, relatif à la taille de la cellule) et sélection des 2 distances les plus grandes, on dispose de bons candidats pour tracer une séparation entre les noyaux.
Grâce à ce post-traitement nous réussissons à améliorer la détection des noyaux avec un score qui passe de 0.32 à 0.34, soit un passage du top 39% au top 34% dans le classement.
Après ces progrès encourageants, nous explorons tout de même une nouvelle piste : le Mask R-CNN
4. L’artillerie lourde : Le Mask R-CNN
Le Mask R-CNN est l’aboutissement de l’évolution de plusieurs architectures de réseaux de neurones. Plus précisément, il s’agit de l’assemblage d’un RCNN (Faster R-CNN) , lui même composé de plusieurs CNNs, ainsi que de couches de convolutions supplémentaires spécialisées dans la segmentation de l’image (cf figure 11).
Mais avant tout revenons sur l’évolution des CNN, dont des mutations successives ont permis d’en améliorer les performances et en élargir les usages.
Le R-CNN
Les premiers CNNs avaient donc comme objectif la classification de l’image. Afin de pouvoir faire de la détection, c’est-à-dire afin de localiser un objet dans une image, une approche naïve et coûteuse consiste à faire glisser une fenêtre dans l’image et interroger notre CNN sur ce qu’il y reconnaît. C’est pour s’affranchir de la lourdeur de cette méthode qu’a été proposé le R-CNN, qui intègre un réseau de proposition de région. Il s’agit d’une première étape qui suggère un certain nombre de régions d’intérêts (ROIs pour Region Of Interest) qui font ensuite l’objet d’une classification (cf figure 10). Cela permet une détection plus rapide.
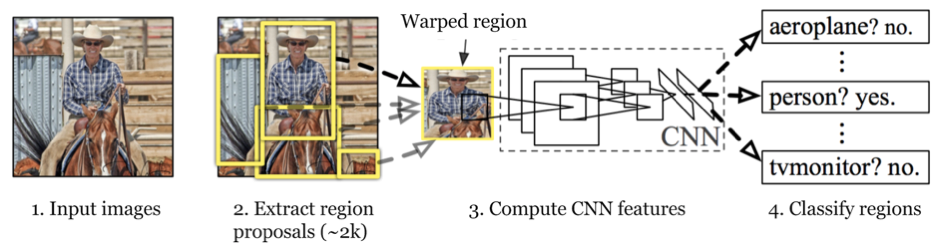
Figure 10 : Schéma explicatif du R-CNN
Ont suivi plusieurs améliorations du R-CNN. Les principales consistent à faire partager au maximum les calculs effectués sur les images aux différentes parties du réseau. Ainsi, une partie des features extraites de l’image entière peuvent être exploitées à la fois pour la proposition des régions, pour la classification et la régression sur la position des box. Cette idée est à l’origine des algorithmes fast R-CNN et faster R-CNN.
Utiliser au maximum les différents niveaux de features du réseau permet d’aller encore plus loin : en ajoutant une courte branche de convolution aux étapes intermédiaires du réseau, on peut en extraire une information de segmentation pixel par pixel. C’est le principe du mask R-CNN
Le Mask R-CNN
Le point clé du Mask R-CNN est de découpler les tâches de classification et de prédiction du masque au niveau du pixel. Sur la base du Faster R-CNN, une troisième branche (branche masque) est ajoutée pour prédire un masque d’objet en parallèle avec les branches existantes pour la classification et la localisation. Cette branche ‘masque’ est un petit réseau entièrement connecté appliqué à chaque RoI et prédisant un masque de segmentation de pixel à pixel (cf Figure 11).
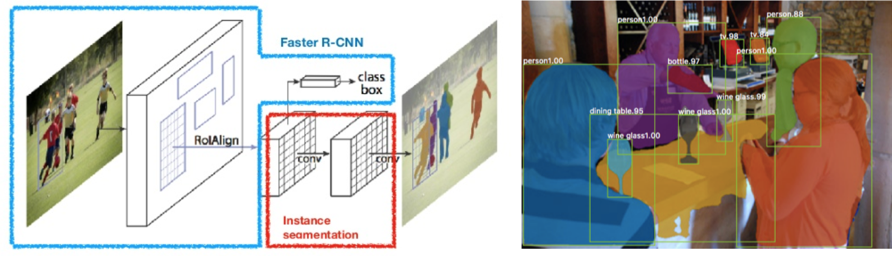
Figure 11 : Principe et exemple du Mask R-CNN.
Étant donnée la profondeur du réseau et le temps nécessaire à le construire de zéro, nous nous sommes appuyés sur une implémentation existante en Tensorflow et Keras, disponible sur le repo ici. Il fait même mention de l’application aux noyaux de cellule depuis la fin du challenge Kaggle !
Le dataset du challenge possédait certaines particularités à prendre en compte dans l’adaptation du modèle. Une seule classe d’objet, la classe noyau, était à détecter (à distinguer de la classe background). Mais les noyaux peuvent avoir des aspects très différents d’une image à l’autre, tous ne provenant pas du même type de cellule. De même, le procédé d’imagerie médicale à l’origine des images du dataset n’était pas toujours le même.
Afin de faire de tourner cette machine de guerre qu’est le Mask R-CNN en un temps raisonnable, nous avons utilisé notre Qbox dont la configuration est donnée plus haut. Elle convenait parfaitement à cet usage, et après l’installation des différentes briques logicielles nécessaire (à savoir driver Nvidia / Cuda / Cudnn / Tensorflow / Keras dont les compatibilités mutuelles peuvent s’avérer frustrantes) nous disposions de quoi entraîner nos modèles.
Vint alors l’heure de paramétrer le réseau ! La limite de la ram de la carte graphique nous a poussés à utiliser le batch-size maximum qui ne causait pas une vilaine erreur OOM (Out Of memory), sans craindre qu’une taille de batch trop grande ne lisse trop l’apprentissage. Malheureusement, le nombre important de masques par image (chacun correspondant à un noyau) nous a contraint à réduire leur taille pour l’entraînement à une centaine de pixels de côté, faisant disparaître les noyaux les plus petits qui ne pouvaient plus être résolus. Différents essais de learning rate et de sa réduction au cours de l’apprentissage, combinés avec un suivi par tensorboard, nous ont permis d’adapter la convergence de notre modèle à nos contraintes de temps.
Stratégies d’apprentissage
Afin de mieux converger et en un temps plus court, nous avons adopté deux stratégies : le transfert learning et la réduction dynamique du learning rate.
Le transfert learning consiste à transférer les connaissances acquises d’un modèle lors de la résolution d’un problème généraliste à un problème différent, plus spécifique mais connexe. Par exemple, les connaissances acquises en apprenant à reconnaître les voitures pourraient s’appliquer lorsqu’on essaie de reconnaître les camions. Cela consiste concrètement à transférer les poids d’un réseau à un autre.
Nous sommes partis des poids du réseaux Resnet50 entraîné sur le dataset ImageNet à classifier 1000 catégories d’objets.
Dans un premier temps nous avons réalisé 20 epochs d’apprentissage en gelant l’ensemble des couches excepté les deux dernières (couche entièrement connectée et couche de classification).
Nous avons ensuite réalisé 20 epochs d’apprentissage avec cette fois ci les 4 dernières couches non figées et terminé par 40 epochs avec l’ensemble des couches libres.
Cette approche nous à permis de converger plus rapidement et d’atteindre de meilleures performances qu’avec une approche classique de ré-entraînement de tous les poids.
Grâce au Mask R-CNN et à cette stratégie nous obtenons un score de 0.42, avec un classement dans le Top 13% de Kaggle. Nous sommes donc à quelques places du fameux Top 10% !
Pour tenter d’y parvenir nous avons mis en oeuvre une deuxième stratégie : Le learning rate dynamique.
Pour faire simple, dans les réseaux de neurone le learning rate est le paramètre qui vas contrôler le pas de convergence (cf figure 12). Un pas de convergence trop faible à l’avantage d’explorer précisément l’espace de convergence, en revanche le modèle nécessite plus de temps pour converger et les risques d’être bloqué dans un minimum local sont importants. Un grand pas de convergence a l’avantage d’accélérer la convergence mais n’explore pas l’espace de convergence de manière optimale et les risques de “louper” le minimum global sont importants.
L’idée d’avoir un learning rate dynamique est de pouvoir optimiser le learning rate en fonction de l’état de convergence du modèle.
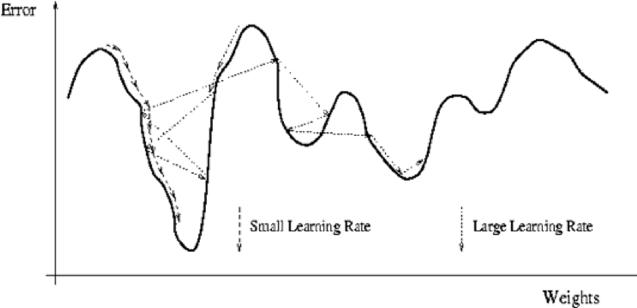
Figure 12 : Schéma explicatif de l’utilisation du learning rate
Finalement, une quinzaine d’heure et une centaines d’épochs plus tard, le modèle est entraîné ! Grâce à cette stratégie nous obtenons un score de 0.44 soit un classement dans le Top10%.
La prédiction est très précise sur les différents types d’images (cf figure 13). La grande force du Mask R-CNN est de pouvoir distinguer les noyaux qui se chevauchent.
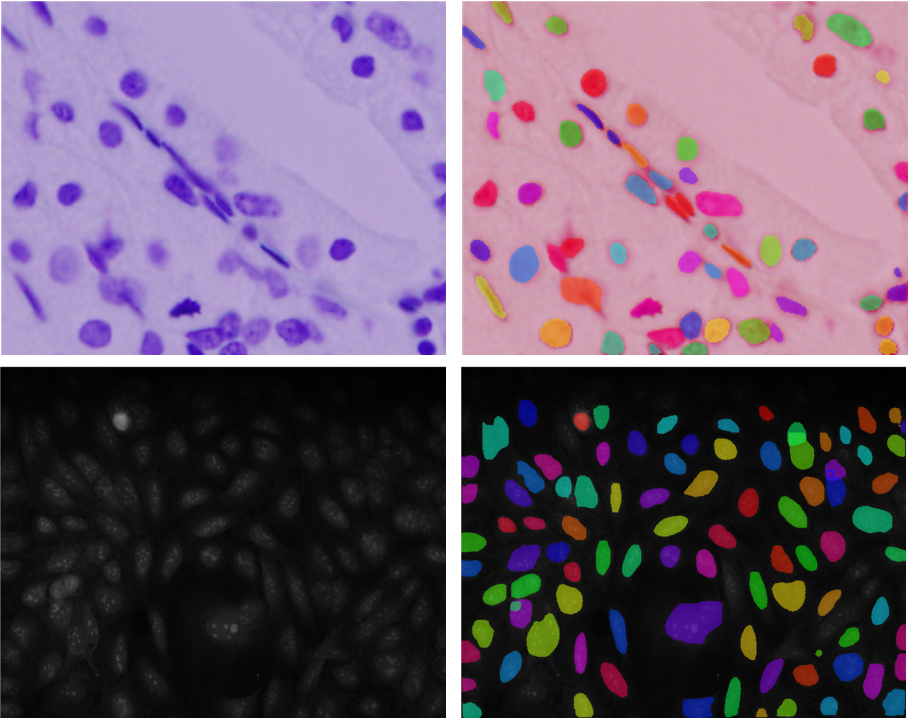
Figure 13: Exemple de détection sur deux image du dataset de kaggle.
Conclusion
Durant ce challenge nous avons donc testé trois approches différentes de complexité croissante:
L’histogramme des couleurs nous a permis de nous échauffer en cernant toutes les problématiques liée aux données et à la cible.
Le modèle U-Net, particulièrement adapté à ce genre de problématique, nous a permis d’avoir une première itération sur nos résultats. Il est simple à prendre en main mais nécessite du post-traitement.
Le Mask R-CNN est quand à lui le candidat idéal pour ce genre de challenge, il nécessite peu de post-traitement, il est en revanche gourmand en ressources de calcul.
✍Écrit par Yassine Ghouzam et Pablo Valverde
Références :
https://www.kaggle.com/c/data-science-bowl-2018
https://arxiv.org/pdf/1505.04597.pdf
https://arxiv.org/pdf/1311.2524.pdf
https://arxiv.org/pdf/1504.08083.pdf
https://arxiv.org/pdf/1504.08083.pdfhttps://arxiv.org/pdf/1504.08083.pdf
https://arxiv.org/pdf/1703.06870.pdf
https://www.tensorflow.org/programmers_guide/summaries_and_tensorboard

